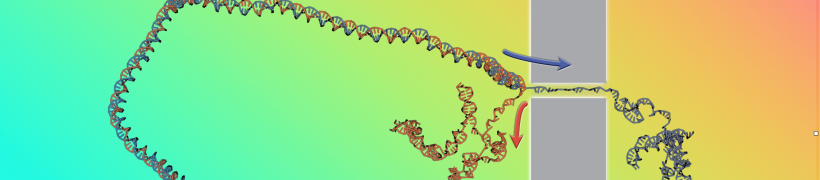
Ricostruire con precisione come le parti di una molecola sono legate tra loro conoscendo solo come la molecola si comporta quando viene deformata o distrutta. È stata questa la sfida affrontata da una ricerca guidata da Cristian Micheletti della SISSA e da poco pubblicata su Physical Review Letters. In particolare, gli scienziati hanno studiando l'apertura di una doppia elica di DNA mentre viene trascinata rapidamente attraverso un nanoporo, ricostruendo proprietà termodinamiche fondamentali del DNA dalla sola velocità del processo.
Il trascinamento di polimeri attraverso nanopori è stato a lungo studiato per le sue complesse proprietà oltre che per la sua rilevanza pratica in molti ambiti, tra cui il sequenziamento del genoma. Quest’ultimo si basa precisamente sul forzare il DNA a passare attraverso un poro così stretto che solo uno dei due filamenti della doppia elica possa entrarvi, mentre l'altro filamento rimarrà indietro. Il DNA, trascinato nel nanoporo da un campo elettrico sarà costretto ad aprirsi e svolgersi, un effetto noto come “unzipping”.
Il team di ricerca, che comprende anche Antonio Suma dell’Università di Bari, primo autore, e Vincenzo Carnevale della Temple University, ha usato un cluster di computer per simulare il processo a diverse intensità di tiraggio conservando traccia della velocità di unzipping del DNA, un tipo di dato direttamente accessibile negli esperimenti ma finora poco studiato.
Utilizzando modelli teorici e matematici elaborati in precedenza, i ricercatori sono stati in grado di lavorare “all’indietro”, usando le informazioni sulla velocità per recuperare in modo accurato la termodinamica di formazione e rottura della doppia elica.

